纳升高通量 PCR
简介
纳升高通量 PCR 是指将高通量的芯片技术与 PCR 技术结合在一起产生的一种新的方法,商用的 TaqMan® OpenArray™系统已将反应体系降至纳升,这对稀少样品的检测极为有利,并且在一张芯片上可以完成多达 3072 个反应,可以进行终点定量,也可以进行实时定量。
原理
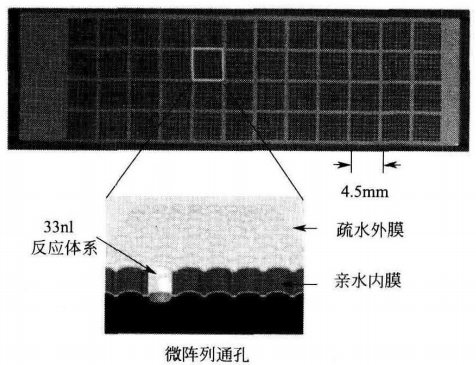
TaqMan® OpenArray™系统的基本原理是是在一块薄的不锈钢电极板(25 mmX75 mmX0. 3 mm)上以 48 个子阵,每个子阵 64 个通 孔的形式排列 3072 个通孔,每个子阵的间距为 4.5 mm, 与 384 孔板相同。每个通孔的直径和深度均为 300/m, 可容纳 33nl 反应体系。芯片表面经过专利的疏水处理,而通孔内壁是亲水且生物兼容的。液体通过表面张 力留于芯片通孔内,试验所需的引物被预先 脱水固化于通孔内壁。使用时将 TaqMan® OpenArray 384 孔样品平板中的 DNA 样品与 PCR master mix 混合。用自动上样仪将样品混合物加样到 TaqMan® OpenArra 严纳升高通量 PCR 芯片中。将芯片插入装满浸液的封箱工作站中,并用封箱胶水密封。在对应的热循环仪上进行循环,用 NT 成像仪成像,并分析数据。图解示意如下。
用途
纳升高通量 PCR 芯片所需样品量小,仅 33nl, 但同批处理的样品数量大,可达 3072 个样 品,适用于大样本而且量稀少的样品的处理。另外,该芯片除了可以对 DNA 样品进行处理,还 可对 RNA 样品进行定量 PCR 口⑶。另外,该方法在菌落的大规模筛选中也可应用⑷。
材料与仪器
器材:
热循环仪(推荐 MJ lower PTC-200); NT 成像仪(Bio Eove Inc.); TaqMan® OpenArrayW 自动上样仪。
试剂:
TaqMan® OpenArray™平板芯片;TaqMan® OpenArray™ 384 孔样品平板(内含待检样 品);PCR Master Mix 含 0. 5% 甘油,1 % Pluronic F38(BASF)和 50ng/ul BSA;封箱胶水(环氧树脂);浸液。
步骤
以 48 个血液样本的 SNP(single nucleotide polymorphism, 单核苷酸多态现象)分型为例,纳升高通量 PCR 的基本过程可分为如下几步:
①利用试剂盒 QIAamp DNA Blood Mini Kit(Qiagen, Valencia, CA)提取分离血液样本(384 孔板中)的 DNA, -20℃ 冻存或将微平板置于冰上。
②以唯一的数字或字母命名微平板的每一个孔,使 SNP 分型软件可以追踪每一个从微平板转移至纳升平板的样品。
③将含有 DNA 样品的微平板室温解冻,1000r/min 离心 1 min, 冰上放置。
④吸取 211ul PCR master mix(10% 多余量)。
⑤在微平板的 48 个孔中,每个孔加入 4ul PCR master mixD
⑥每个 DNA 样品吸取分别与 4ul PCR master mix 吹吸混匀。
⑦用铝箔盖住含有 DNA 样品的微平板,1000r/min 离心 1 min 以去除气泡。冰上使样品冷却。
⑧用钳状骨针铝箔,暴露要转移至纳升高通量 PCR 芯片的 48 个样品,自动上样仪将样品加至芯片透孔中。
⑨将芯片插入装满浸液(一种与 PCR 反应体系不混溶的全氟化液体)的封箱工作站中,以 UV 照射环氧树脂密封,防止反应体系在热循环中挥发。
⑩将密封好的芯片放入热循环仪,设定程序如下:91°C 预变性 10 min, 然后以 51°C 23s、 53. 5°C 30s. 54.5°C13s、97°C 22s、92°C 7s 进行 50 个循环。循环结束后保持在 20°C 直至芯片被 成像处理。
⑪用 NT 成像仪成像,并分析数据。
注意事项
①PCR master mix 的体积应多余 10% , 防止吸头的误差。
②用 OpenArray 自动上样仪将样品混合物上样到 TaqMan® 0 卩 2 話口汕即平板芯片中时, 芯片与吸头末端形成液体三明治结构。但是液体不会沾湿芯 片表面,因为芯片的表面经过疏水化处理,液体会迅速、 精确地进入每一个经过亲水化处理的透孔。
③在 SNP 分型中,DNA 的量和质量会影响准确率。 如图 8-6 所示,在三组试验中(分别用三角、圆圈和方框 代表),DNA 的质量不同,每个反应中 DNA 的浓度在 0.01〜Ing 之间,可以看出不同浓度的准确率各有不同。 但是有些反应,如三角代表的一组,在 DNA 的浓度低至 0. Olng 时准确率仍高达 90%;而有些反应,如方框代表的 一组,在 DNA 的浓度达到 0. 15ng 时,准确率才能达到 90%, 而小于 0. 15ng 时准确率就会迅速下降,在 0. Olng 时准确率不到 70%。这种准确率的改变为杂合子和纯合子 的分离增加了难度。

④TaqMan 试验的再设计也会增加纳升平板芯片的效果。图 8-7 所示为 TaqMan SNP 分型的 两次试验,图(b)中的试验与图(a)相比在探针的 5』末端添加了一个单核苷酸。这个改动大大提 高了基因分型的效果。
30000r 30000r

0 . . — | o' <strong~undefined< strong="" style="box-sizing: border-box; border: 0px solid; --tw-border-spacing-x:0; --tw-border-spacing-y:0; --tw-translate-x:0; --tw-translate-y:0; --tw-rotate:0; --tw-skew-x:0; --tw-skew-y:0; --tw-scale-x:1; --tw-scale-y:1; --tw-pan-x: ; --tw-pan-y: ; --tw-pinch-zoom: ; --tw-scroll-snap-strictness:proximity; --tw-gradient-from-position: ; --tw-gradient-via-position: ; --tw-gradient-to-position: ; --tw-ordinal: ; --tw-slashed-zero: ; --tw-numeric-figure: ; --tw-numeric-spacing: ; --tw-numeric-fraction: ; --tw-ring-inset: ; --tw-ring-offset-width:0px; --tw-ring-offset-color:#fff; --tw-ring-color:rgba(59,130,246,0.5); --tw-ring-offset-shadow:0 0 #0000; --tw-ring-shadow:0 0 #0000; --tw-shadow:0 0 #0000; --tw-shadow-colored:0 0 #0000; --tw-blur: ; --tw-brightness: ; --tw-contrast: ; --tw-grayscale: ; --tw-hue-rotate: ; --tw-invert: ; --tw-saturate: ; --tw-sepia: ; --tw-drop-shadow: ; --tw-backdrop-blur: ; --tw-backdrop-brightness: ; --tw-backdrop-contrast: ; --tw-backdrop-grayscale: ; --tw-backdrop-hue-rotate: ; --tw-backdrop-invert: ; --tw-backdrop-opacity: ; --tw-backdrop-saturate: ; --tw-backdrop-sepia: ; line-height: 1.625rem !important;">■ 1— | |||
0 4000 8000 12000 | 0 | 4000 | 8000 | 12000 |
' VIC荧光值 | VIC荧光值 | |||
| (a) | (b) |
图 8-7 通过引物探针的再设计增加基因分型的有效性
⑤引物可以使用软件 Primer3 设计或在引物库中筛选,引物的退火温度应在 60〜65C, 采 用退火温度最接近的引物对。引物序列的长度应在 18〜25 个核苷酸,(G+C)含量在 30%〜 60%。每一对引物都经过磷酸化处理,通过离子键与芯片内壁表面结合,直到 PCR 反应的开始。
相关文章
- RT-PCR、Western blot和ELISA三者的区别
- PCR反应五要素
- 原位聚合酶链式反应(in situ PCR)和原位反转录聚合酶链式反应(in situ RT-PCR)操作规程
- A sensitive quantification of HHV-6B by real-time PCR
- PCR标准反应体系
- Validation of RNAi by Real Time PCR
- Site-Directed Mutagenesis and Gene Fusion by Megaprimer PCR
- 逆转录-聚合酶链反应 (Reverse Transcription-Polymerase Chain Reaction,RT-PCR
- 锚定PCR
- Real-Time PCR Fluorescent Chemistries




