SSR标记介绍
什么是SSR?
短串联重复序列(Short tandem repeat, STR)=微卫星DNA( microsatellite DNA) = 简单重复序列(simple sequence repeat , SSR)
长度多态性遗传标记,一般由 2-6个碱基的DNA重复序列组成,其长度多态性来源于重复单位拷贝数的个体差异。
STR广泛存在于基因组中,平均每6-10 kb就存在一个STR基因座。人类基因组内已发现了七千个以上的STR位点。
(所以并不是非酋抽不到的SSR……哦现在抽不到的是UR……玄不改命,氪不救非!)
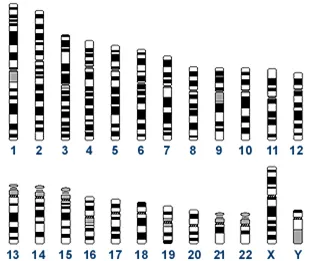
法医学应用的STR在染色体上的位置
23 CODIS Core STR Loci
微卫星DNA的特点
Ø数据大、分布广且均匀
Ø具有极高的个体特异性
Ø多态信息含量高
Ø共显性遗传
(逐渐开始做实验用的专业知识……)
适用于DNA指纹的SSR基因座的选择
较高的个体识别率,通常>0.9,观测杂合度>0.7
在染色体上定位相对较远,确保无连锁
低stutter产物、低突变率
与其他遗传标记复合检测时易于得到结果,且具有重现性
鉴定的位点越多,判错的可能性越小,个体识别能力越来越强
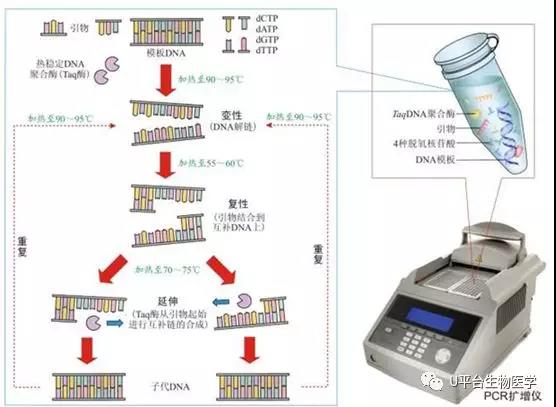
SSR技术原理
根据微卫星DNA两端序列的保守性,设计一对特异性的引物,利用PCR技术扩增每个位点的微卫星DNA序列,以检测分析微卫星序列的多态性,从而进行相关的遗传学分析。
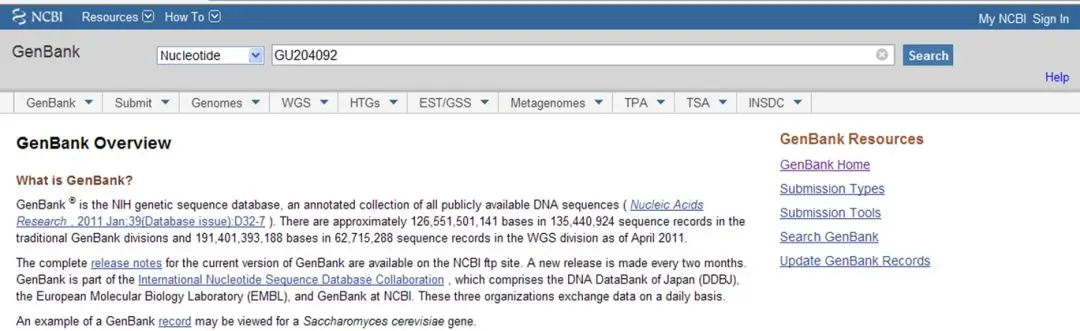
SSR位点确定-Genbank搜索序列
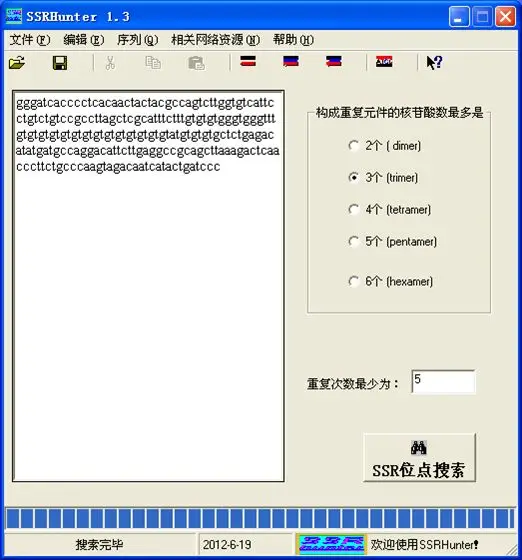
SSR位点确定-SSRHunter1.3软件
标准化是DNA指纹库建立及应用于品种鉴定实践的前提和基础,DNA指纹库能否应用的关键是不同实验室对同一样本测试的基因型必须完全一致,数据具有可比性。
以下是目前人类DNA指纹库和我国玉米DNA之文库的建立情况:
人类DNA指纹库
美国CODIS系统:13个STR标记
英国:6个STR标记
欧洲:7个STR 标记
我国玉米DNA指纹库
承担农业部国家区试玉米DNA指纹鉴定方法标准及管理办法制定
承担农业部品种权保护玉米DNA指纹鉴定方法标准制定
参与国际UPOV的关于“分子标记选择与指纹库构建”的BMT指南的修订