设计自定义的sgRNA文库
简介
尽管每个 sgRNA 文库都是为特定目的而计算设计的,但基本的设计过程在文库之间是一致的。
首先,根据已知的 sgRNA 靶向规则(如保守外显子 5' 用于基因敲除,转录起始位点的上游或下游分别用于转录激活或抑制),确定感兴趣的靶向 sgRNA 文库的基因组区域。
其次,根据以下四个标准识别和选择 Cas9 同源特异性前间区序列邻近基序 (PAM) 的所有可能的sgRNA靶标,要求:① 脱靶活性最小化;② 靶标活性最大化:③ 避免同聚体延伸(如 AAAA、 GGGG);④ 一定的 GC 含量。
材料与仪器
器材:python 脚本
步骤
设计自定义的 sgRNA 文库的基本过程可分为如下几步:
(一)生成针对自定义基因组坐标的文库
A 文库一代 python 脚本的安装要求。python 脚本 design_jibrary.py 会生成—组靶向指定基因组坐标的 sgRNA 。安装 python 2.7, twobitreader, biopython 和 seqmapo 对于 seqmap ,安装适用于所有平台的版本1.0.13源代码,并使用 g++ - 03 -m64 -0 seqmap match.cpp 进行编译。将 seqmap 放在与 python 脚本 design_jibrary.py 相同的文件夹中。
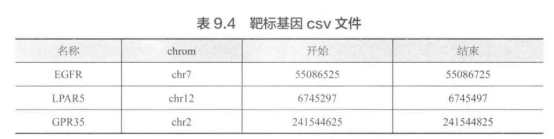
B输入靶标基因组坐标进行文库设计。一旦确定了自定义 sgRNA 库的一组基因和坐标,从左到右在每一列中准备一个包含基因名称、染色体、目 标区域开始和結束的靶标基因 csv 文件。紹标基因 CSV 文件应该包含标校 名称、 chrom、开始和结束。提供的 python 脚本将识别靶向基因组区域内 每个基因的潜在 sgRNA ,如靶标基因 csv 文件中所述。参考表 9.4 中的示例 输入文件。
C 设计自定义文库。从 UCSC genome Browser (http://hgdownload.cse.ucsc. edu/)下载靶标基因坐标对应的基因组 2 bit 文件。基因组 2 bit 文件将用于构 建一个基于每个间隔序列和基因组中相似序列之间的不匹配位置和分布的脱 靶分数数据库。对于靶标基因 csv 文件中的每个区域,python 脚本将识别潜 在的 sgRNA ,并使用此数据库作为自定义库,选择指定数量的 sgRNA 和更少 的潜在脱靶位点。要设计自定义库,运行 python design_library.py ,并使用以下可选参数(表 9.5)。

在针对大型基因组区域(> 50 kb)设计 sgRNA 时,建议将靶标基因 csv 文件拆分成包含不同靶标基因子集的几个文件,以并行运行文库设计过程, 节约运行时间。在 design_jibrary 运行后,将目标指定基因组坐标的间隔序列 写入输出 csv 文件。当设计一个新的自定义库,且目标是与以前的自定义库 相同的基因组区域时,使用以前构建的脱靶数据库可以显著减少脚本执行时 间。如果指定 -gecko 或 -sam ,则包含用于合成的间隔和各自侧翼序列的整个 Oligo 库序列将位于最后一列。
(二)从现有库生成目标库
A输入靶标基因进行文库设计。python 脚本 design targeted library.py 从针对特定基因组的现有库中提取 sgRNA 间隔子。安装 python 2.7(https:// www.python.org/downloads/)。确定了靶向筛选的一组基因后,准备一个csv 文件,其中包含靶标基因的名称,每行对应一个基因。为带注释的基因组级 的文库准备另一个 csv 文件,第一列是每个基因的名称,第二列是各自的间 隔序列。每一行包含一个不同的间隔序列。靶标基因文件中的基因名称应该 与注释库文件中的名称釆用相同的格式。
B 设计有针对性的自定义文库。使用以下可选参数(表 9.6)运行 python design_targeted_library.py ,从基因组规模文库中分离与靶标基因相对应的间隔子集。

在 design_targeted_library 运行后,将靶基因的间隔子集写入输出 csv 文 件。如果指定 -gecko 或 -sam ,则包含用于合成间隔和各自侧翼序列的整个 Oligo 库序列将位于最后一列。
(三)通过DNA合成平台(例如 Twist Bioscience 或 Custom Array)合成 Oligo 文库为阵列作为混合文库。合成通常需要 2~4 周,具体取决于 Oligo 文库的大小。用封口膜封口并在 -20 ℃下储存寡核昔酸。