基于 PCR 的长 DNA 序列准确合成法
简介
基于 PCR 的长 DNA 序列准确合成的方法(PCR-based accurate synthesis, PAS)相对简便(只需要 2 步 PCR)、快速、准确(每合成 1 000 bp 错配 ≤ 1)和廉价,已经成功合成了大量的基因,包括含高(G + C)含量、重复序列和复杂的二级结构的基因。
Xiong 等人应用 PAS 法合成了枯草芽弛杆菌次次操纵子的一个 12 kb 的 DNA 片段。运用 PAS 法设计合成 DNA 序列可以优化密码子和(G + C)含量,将不需要的限制性内切酶位点和二级结构移开。
原理
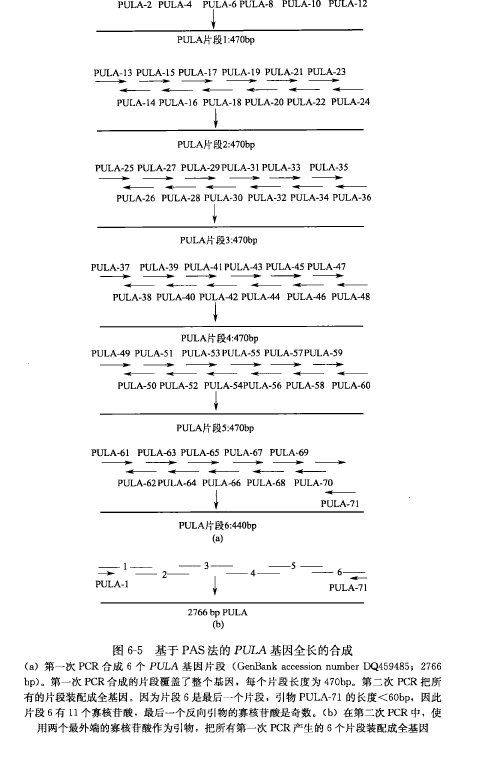
基于 PCR 的长 DNA 序列准确合成法的基本原理是。根据目的 DNA 序列,设计并化学合成与相邻的核苷酸有 21 bp 重叠的长 60 bp 的寡核苷酸。为了降低化学合成中的错误,所有的寡核苷酸应该用 PAGE 进行纯化。一组 12 个彼此相邻的 60 bp 的寡核苷酸通过 PCR 合成 400~500 bp 长度的 DNA 片段【图 6-5 (a)】;大多数的 5' 正向寡核苷酸和 3' 反向寡核苷酸(外部寡核苷酸)以保留的 10 个内部寡核背酸作为模板,采用高保真的 DNA 聚合酶(比如 Pfu,每 1 000 bp 小于 1 个错误)。
由于这 2 个外部寡核苷酸的浓度是内部寡核苷酸的 20 倍,大多数的 PCR 产物应该是大约 400~500 bp 的 DNA 片段。第二步 PCR 时,应用第一步 PCR 合成的所有 400~500 bp 的 DNA 片段作为模板,合成全长 DNA 序列【图 6-5(b)】。在这步 PCR 中,应该使用产生长的准确的 DNA 片段的 DNA 聚合酶 (例如,焦硼酸钠 DNA 聚合酶,能够合成达到 10 kb 长的片段,具有与 Pfu 相似的低错误率)。最后,通过 DNA 序列分析鉴定合成的基因序列,用重叠延伸 PCR 技术校正序列中的误差。
材料与仪器
器材:
① EP 管;
② PCR (GeneAmp PCR System 9600);
③ ABI PRISM 310 生物分析仪等。
试剂:
① BigDye Terminator v3.1 循环测序法试剂盒;
② 寡核苷酸混合物。
步骤
基于 PCR 的长 DNA 序列准确合成法的基本过程可分为如下几步:
一、第一步 PCR 400~500 bp DNA 片段的合成
1、将一组 12 个彼此相邻的 60 bp 的寡核苷酸合成一个 400~500 bp 的 DNA 片段(图 6-5)。在整个过程中,所有的反应试剂应该贮存和在冰上处理。如长为 2 766 bp 的 PULA 基因,所有合成的寡核苷酸被分成 6 组,合成每个 DNA 片段[见图 6-5(a)]。

2、对于每个组,将 10 个内部引物混合:每个引物取 1 μL(30/μmol/L)加入到一个 EP 管里,轻轻混匀。阳性对照为「已知模板」,其中有 10 个内部寡核甘酸被替换为全长基因的适当序列的;阴性对照为「无模板」,即用水代替了内部寡核苷酸;同时,采用加入每对外部引物中的一个引物即「单个引物」作为阴性对照。以 PULA 基因为例,管 A1,A2,A3,A4,A5 和 A6 自包含了相应的 10 个内部寡核苷酸。
3、取出寡核苷酸混合物 0.5 μL 加入到 500 μL 的 PCR 离心管中。如 PULA 基因,从管 A1,A2,A3,A4,A5 和 A6 里取出 0.5 μL 到相应的管 B1,B2,B3,B4,B5 和 B6[图 6-5(a)]。
4、加入每对外部引物(30 pmol)1 μL,每管包含合成一个 400~500 bp 的 DNA 片段的所有的 12 个寡核苷酸。
5、然后在每个管里(包含总量 2.5 μL 的 12 个寡核甘酸混合物)加入下面的 PCR 反应物:

将内容物在管里轻弹混合并离心。
6、在下列条件下进行 PCR(GeneAmp PCR System 9 600):

7、从每管 PCR 产物取出 10%(5 μL)进行琼脂糖凝胶(1.0%)电泳,150 V,20 min;对 PCR 产物进行定量,从第一步 PCR 得到的单链 DNA 产物至少 100~200 ng, 以确保从第二步 PCR 得到高质量的产物。在第一步 PCR 看到有多重条带时,靶基因应进行凝胶纯化。为了合成长 DNA 序列(例如,4 kb),在收集全长 DNA 前先合成 2 kb 的中间产物,克隆和测定这些中间物,并对它们进行误差校正后再继续下步实验。
二、第二步 PCR 装配全长 DNA 序列
8、对第 7 步的所有 PCR 产物,从每个产物取 1 列(相当于 50 ng DNA)混合到 500 μL 的 EP 管里(图 6-5)。注意事项:从第一步 PCR 的每个产物里取出大约相等的量加入,以获得高质量的全长 DNA 进行第二步 PCR。
9、然后取出 1 μL 混合物加入到一个 500 μL 的 PCR 管里。
10、加入全长 DNA 的最外部的 2 个引物(30 pmol),如 PULA 基因,加入寡核苷酸 1 和寡核苷酸 71[图 5-6(b)]。同时加入以下 PCR 反应物进行 PCR:

11、PCR(GeneAmp PCR System 9 600)条件为:以「已知模板」作为阳性对照,即以全长基因代替第一步 PCR 得到的 DNA 片段作为模板;「无模板」即用水代替做模板和「单引物」作为阴性对照。
12、将得到的全部 PCR 产物在 1.0% 的琼脂糖凝胶上进行琼脂糖凝胶电泳,电压 150 V, 时间 20 min, 进行凝胶纯化。应该获得大小正确的一条干净、单一的条带。第二步 PCR 得到的 DNA 片段产物应该进行凝胶分离纯化,以使后面的克隆更加容易进行。注意事项:应对合成产物的准确度进行验证及错误校正。
13、对得到的 PCR 产物克隆到任何的载体里,进行 DNA 序列分析。注意事项:焦硼酸钠聚合酶得到的 PCR 产物为平头末端,如果使用的是 TA PCR 克隆试剂盒,那么必须加入 3'-氨基嘌呤。
14、为了保证合成的 DNA 不存在错误,使用 BigDye Terminator v3.1 循环测序法试剂盒和 ABI PRISM 310 生物分析仪进行测序。对于有害的错误,可以利用 OE-PCR 校正[以 PULA 基因为例,见图 6-6(a)]。PAS 终产物的错误主要有两个原因:化学合成寡核苷酸时的错误和从 PCR 造成的产物错误。当前用来合成寡核苷酸的方法,尤其是利用多频合成器进行长寡核苷酸高通量的合成时,常常会引起序列过早终止或者内部缺失。如果使用未纯化的寡核苷酸,错误率将高达每合成 1 000 bp 有 5~10 个错误。而使用 PAGE 纯化的寡核苷酸,错误率将降低到每合成 1 000 bp 约有 1 个。因此,用 PAGE 纯化寡核苷酸是很必要的。虽然使用 PAGE 纯化可以去除不正确长度的寡核苷酸,但是无法检测并去除碱基置换的错误。
三、OE-PCR 验证
15、为了能够校正每个错误,在反应方案里设计一对精确的补充引物。例如在合成 PULA 基因中,发现了 3 个错误:A 589 G, C 1320 T 和 T 2120 A [图 6-6(a)]。因此化学合成 6 个 DNA 序列正确的新寡核苷酸(长度为 25 bp)覆盖三个有错误的部分。这 6 个寡核苷酸命名为 PULA-M1, PULA-M2, PULA-M3, PULA-M4,PULA-M5 和 PULA-M6[图 6-6(b)]。每对寡核苷酸覆盖一个错误的区域。比如,寡核苷酸 PULA-M1 和 PULA-M2 覆盖了包含错误 A 589 G 的区域[图 6-6(a)]。简短地说,OE-PCR 的第一步使用 PCR 合成的 DNA 片段Ⅰ,Ⅱ,Ⅲ和 IV[图 6-6(a)],利用 PAS 方法生产的 PULA DNA 序列作为模板。DNA 片段Ⅰ,Ⅱ,Ⅲ和 IV 应该已校正了 3 个错误。第二步以混合的 DNA 片段Ⅰ,Ⅱ,Ⅲ和 IV 作为模板,PCR 合成完整的 PULA 基因。

16、对 PCR 管进行编号,每个管里加入 100 ng 第二次 PCR 合成的全长 DNA。加入 1 μL(30 pmol)适当的上下游引物到相应的管里。例如 PULA 的合成,对于管 Ⅰ,加入 PULA-1 和 PU- LA-M2[图 6-6(b)]扩增 DNA 片段 Ⅰ[图 6-6(a)]。对于管Ⅱ,加入 PULA-M1 和 PULA-M4[图 6-6(b)]扩增 DNA 片段Ⅱ[图 6-6(a)]。对于管Ⅲ,加入 PULA-M3 和 PULA-M6[图 6-6(b)]扩增 DNA 片段Ⅲ[图 6-6(a)]。对于管 IV,加入 PULA-M5 和 PULA-71[图 6-6(b)]扩增 DNA 片段 IV[图 6-6(a)]。
PCR 的条件如下:

17、通过 PAGE 纯化 PCR 产物。
18、混合 PAGE 纯化的 DNA 片段(每个片段 100 ng)。取 1 μL 从第 ⑩ 步得到的每对最外部寡核苷酸作为引物合成全长 DNA(GeneAmp PCR System 9 600)。进行 PCR,条件如下:

19、将得到的全部 PCR 产物在 1.0% 的琼脂糖凝胶上进行琼脂糖凝胶电泳,电压 150 V, 时间 20 min。观察是否得到预期的单一条带,然后进行基因测序。
相关文章
- PCR-SSCP的发展现状
- PCR-SSCP注意的事项
- PCR-SSCP原理和操作步骤
- 内切酶在PCR反应产物中的活性(Activity of Restriction Enzymes in a Primer Extensio
- RT-PCR Analysis--详细的RT-PCR方法
- RT-PCR: The Basics
- Real-Time or Kinetic PCR
- PCR Amplification from Microbial Colonies
- Calculating Concentrations for PCR
- PURIFICATION OF PCR PRODUCTS WITH SEPHAD