降落PCR
原理
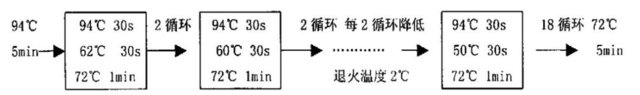
降落 PCR 是 Dn 于 1991 年最早发明的指每一个(或 n 个)循环降低 1(或 nC)退火温度,直至达到一个较低的退火温度,这个温度称为“touchdown”退火温度,然后以此退火温度进行 10 个左右的循环。据统计,正确和非正确退火温度之间的 1 差异将造成 FR 产物量的 4 倍差异,如果相差 5 就会产生 42(1024)倍的优势,因此相对于非正确产物,正确的产物可以得到富集。退火温度起始在高于计算的T值的 15 左右,在接下来的循环中。
退火温度以每次 12 逐渐降低,直到 T 值以下 5 当达到特异的引物模板结合的 T 值时,扩增就会开始。当退火温度降到非特异扩增发生的水平时,特异产物会有一个几何级数的起始优势,在剩余反应中特异产物优先扩增,从而产生单一的占主导地位的扩增产物。这种方法主要用于避免非特异 PCR 产物的出现,尤其是当使用复杂的基因组 DNA 模板,非特异的退火更容易发生时。
降落 PCR 中正确的产物得到富集,原理在于温度的升高提高了 PCR 扩增的特异性,但也提高了引物结合的难度,降低了扩增的效率,因此一开始先用高温扩增,保证扩增的严紧性,待目的基因的主度上升后,降低扩增的温度,提高扩增的效率(此时非特异的位点由于丰度低,无法和特异位点竞争)。但是,降落 PCR 无法改善扩增效率低的问题,一般用于在杂模板中提高扩增特异性。
用途
把降落 PCR 与定量 PCR 结合起来,检测样品中的病原数量,敏感性好,精确度也高在 LarsenHI 等人的研究中,当他们使用退火温度为 50 的常规 PCR 程序时,得到的结果不尽如人意。后来改用了降落 PCR 的方法退火温度由65降到50(2~6个循环每个循环降低 1:7~11 个循环每个循环降低,然后在退火温度为 50 时 35 个循环。结果表明,使用降落 PCR 时,12 个样品全部检测为阳性;而同样条件下,使用常规PCR 程序 12 个样品中只有 7 份是阳性。
在 Seoml 等的工作中,他们试图设计一对引物去同时检测兰科植物中感染很广的两种w# cymbidium mosaic potexvirus(CymMV)Fl odontoglossum ringspot tobamovirus(ORSV)预计扩增 PCR 的片段分别是 534bp(CymMV)和290bp(ORSV),当使用标准 PCR 程序扩增时,退火温度为 55,但是 CymMV 的 534bp 片段的扩增效率远远高于 ORSV 的 290bp 片段 290bp 片段几乎看不见,可能是 CymMV 的这段序列(G+C)含量较高,有较高的退火结合温度。后来他们尝试了降落 PCR 的方法,起始温度分别为 504846 每个循环降低 0.5 共 30 个循环。
结果表明当起始退火温度为 48 时,电泳检测两条带的强度相似,产物比起标准 PCR 程序都有所增加。说明降低起始退火温度,更有利于引物与 ORSv 结合。但当降落 PCR 的起始退火温度降到 46 时,就出现了较多的非特异产物。因此,最后他们选择了起始退火温度为 48 的降落 PCR 降落 PCR 的另一个应用是在确定已知氨基酸序列肽的 DNA 序列。
具体过程如下:
使用两条与已知序列肽两末端可能配对的简并引物,需要知道一段长为 13 个氨基酸的肽段序列,5 和 3 引物各长 18 个碱基(6个氨基酸),两者之间有一个碱基或更长的间隔,进行“touchdownPCR”。由这种方法将获得大量的产物,但因为由肽序列可以知道引物之间的确切距离,所以可以根据产物的大小来选择所需要的产物。
该方法的优点在于可以富集引物与模板正确配对的产物。如果克隆彬蔚和测序几个PCR产物,将可以确定肽的正确编码DNA序列,可用于设计进行杂交的寡核苷酸。该
技术特别适用于由Serlys和Arg(每个有6个密码子)构成的多肽。
材料与仪器
基因样品 引物 去离子水 DNA 聚合酶 缓冲液 dNTP MgSO4(可选) 和 DMSO(可选)PCR
步骤
1.反应体积为 50 ul。
2.人 CD137 胞膜外区基因的 PCR 扩增体系:CD137-pCDNA3 质粒 4 ul,P1,P2各 0.5 umol/L,MgCl2 2 mmol/L,dNTP 0.4 mmol/L,Taq 5 U。
3. hIgG1Fc 片段的 PCR 扩增体系:含人 hIgG1Fc 的 PGEM-T 质粒 4 ul,P3、P4各 0.4 umol/L,MgCl2 2 mmol/L,dNTP 0.4 mmol/L,Taq 5 U。
4. HBVDNA 多聚酶基因的 PCR 扩增体系:
(1)第一轮:HBVDNA 模板 4 ul,P5、P6 各 0.25 umol/L,MgCl2 1.5 mmol/L,dNTP 0.2 mmol/L,Taq 0.8 U。
(2)第二轮:取 5 倍稀释的第一轮 PCR 产物 4 ul 为模板,P7、P8 各0.25 umol/L,MgCl2 1.5 mmol/L,dNTP 0.2 mmol/L,Taq 0.8 U。
5.分别在各灭菌 PCR 管加入上述各反应成分,100℃ 煮沸 5 min,立即冰浴 5 min,加入 TaqDNA 聚合酶,混匀,离心,PCR 程序如下:取 10 ul PCR 扩增产物,在 2% 或 3% 琼脂糖凝胶上电泳,紫外灯下观察扩增产物。
常见问题
1.设置热循环仪程序。下面的时间表适用于所计算的 T 值为 60℃ 的情况。
循环号 变性 复性 延伸
1 、 94℃,1min 66℃,30s 72℃,1min
2 、94℃,1min 65℃,30s 72℃,1min
3 、94℃,1min 64℃,30s 72℃,1min
4 、94℃,1min 63℃,30s 72℃,1min
5 、94℃,1min 62℃,30s 72℃,1min
6 、94℃,1min 61℃,30s 72℃,1min
7 、94℃,1min 60℃,30s 72℃,1min
8 、94℃,1min 59℃,30s 72℃,1min
9 、94℃,lmin 58℃,30s 72℃,1min
10、94℃,1min 57℃,30s 72℃,1min
11、94℃,1min 56℃,30s 72℃,1min
12~27、94℃,1min 55℃,30s 72℃,1min
2.建立一个方案 2 中描述的热启动 PCR。在方案 2 中的步骤 6,执行上面的时间表进行扩增反应。注意总循环数不要超过 30~35,否则会出现非特异性产物和/或引物二聚体。