用 PCR 来制备编码随机肽的双链 DNA 文库实验
材料与仪器限制性内切酶和缓冲液 灭菌双蒸水 Tris-HCl 生物素标记的 PCR 引物链霉抗生物素蛋白磁性珠 琼脂糖凝胶电泳所需的试剂和设备步骤一、材料1.
材料与仪器
限制性内切酶和缓冲液 灭菌双蒸水 Tris-HCl 生物素标记的 PCR 引物
链霉抗生物素蛋白磁性珠 琼脂糖凝胶电泳所需的试剂和设备
链霉抗生物素蛋白磁性珠 琼脂糖凝胶电泳所需的试剂和设备
步骤
一、材料
1. 缓冲液、溶液和试剂
灭菌双蒸水(ddH20)
10 mmol/LTris-HCl,pH8.0
2. 酶和酶缓冲液
限制性内切酶和缓冲液(见第 9 步)
10XVent 缓冲液
VentDNA 聚合酶
3. 核酸和寡核苷酸
生物素标记的 PCR 引物,长度至少 15bp,5'末端进行了生物素修饰(见图 26-1)。
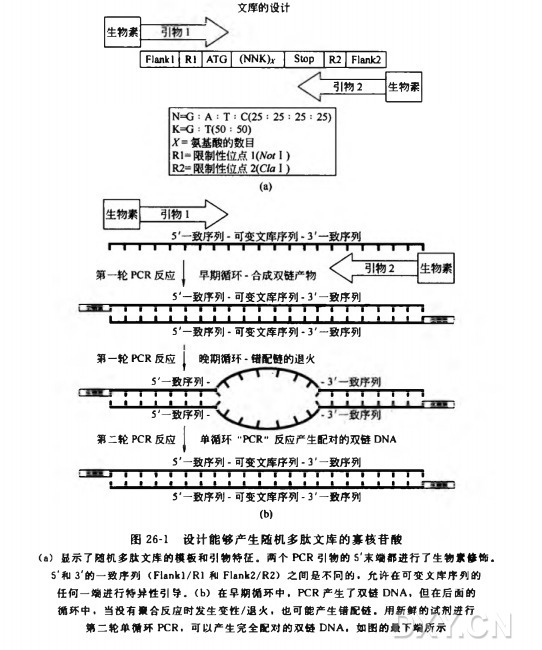
编码 12 个氨基酸的 NNK 库。库的设计如图 26-1(a) 所示。
合成的链含有一个随机多肽库,它们的 3'末端和 5‘末端都加了一个特定的侧翼序列,作为引物结合的位点,并且含有限制性内切酶的识别位点,以方便将文库 DNA 克隆进合适的载体系统。侧翼序列长度至少应该有 15bp,并且设计时应该有 3 个标准:1.它们应该是有效的 PCR 引物结合位点;2.它们应该含有后续克隆步骤所需的限制性酶切位点(可选:限制性酶切位点可以位于 PCR 引物 5’端与文库寡核苷酸的重叠区,而不要位于文库寡核苷酸内部),3.因为一端或两端的便翼 DNA 序列有可能在肽库中代表氨基酸,它们应该不包含影响文库功能的密码子。在合成文库的编码区过程中,每个密码子的前两个位点中,4 种核苷酸的含量是相等的,而在第三个位点,G+T 的比例是 1:1[图 26-1(a)]。在第三个位点不使用 A 和C 核苷酸,减少了形成终止密码子的可能,但仍允许编码 20 种氨基酸。
dNTP(各 20 mmol/L)
4. 其他
琼脂糖凝胶电泳所需的试剂和设备,包括溴化乙锭 (见第 11 步)
QIAprep Miniprep silica column(QIAGEN)
链霉抗生物素蛋白磁性珠(Dynal)
二、方法
1. 第一轮聚合反应:文库的 PCR 扩增
(1)进行下列反应,将单链的合成寡核苷酸,扩增为有效的构建文库的起始材料。下面所给的是 100ul 体系的用量。要构建大规模的库,准备 1 ml 的反应物,并将它们平分到 10个独立的 PCR 管中。
10pmol 的文库寡核苷酸
2U 的 Vent DNA 聚合酶
生物素标记的 PCR 引物,各 100pmol
10ul 的 10XVent 缓冲液
dATP、dCTP、dTTP 和 dGTP,各 1ul
用 ddH2O 定容至 100ul
(2)在一个热循环仪中,94°C 将混合物预热 20s。
(3)按下面的参数进行 30 轮 PCR 循环。
94°C10s
55°C10s(或用引物的合适退火温度)
72°C15s
(4)冷却至 4°C。
2. 第二轮聚合反应:合成配对的双链文库 DNA.
(5)在每 100ul 第一轮反应产物中,加入 10ul 的 10X 反应缓冲液、100pmol 的各引物、2U 的 Vent DNA 聚合酶。
(6)进行一个单轮的循环:94°C 30s,55°C 30s,72°C 15s。
(7)冷却至 4°C。
(8)用 QIAprep 的柱子纯化第二轮扩增产物,并用 10mmol/L Tris(pH8.0) 洗脱。
(9)用合适的内切酶消化洗脱产物(图 26-2,第 1 泳道),然后用乙醇沉淀 DNA, 并用ddH2O 溶解(Sambrook and Russell2001)。经过这一消化反应,生物素标记的 DNA 末端将从文库的核心释放出来。
(10)用链霉抗生物素蛋白磁性珠去掉生物素标记的 DNA 末端(Korn et al.1992)。这一步骤将增加目的连接产物的产量,因为这些末端能够与文库插入片段或表达载体相连接。链霉抗生物素蛋白的亲和步骤,同时能去掉任何没有酶切或部分酶切的 PCR 产物(图 26-2,第 3 泳道和第 4 泳道)。
(11)在 4% 的琼脂糖凝胶上,分析反应中间产物和终产物。终产物(图 26-2, 第 4 泳道)是高纯度且高度复杂的双链文库 DNA,有突出的末端,可以直接连接到合适的表达系统上。

1. 缓冲液、溶液和试剂
灭菌双蒸水(ddH20)
10 mmol/LTris-HCl,pH8.0
2. 酶和酶缓冲液
限制性内切酶和缓冲液(见第 9 步)
10XVent 缓冲液
VentDNA 聚合酶
3. 核酸和寡核苷酸
生物素标记的 PCR 引物,长度至少 15bp,5'末端进行了生物素修饰(见图 26-1)。

编码 12 个氨基酸的 NNK 库。库的设计如图 26-1(a) 所示。
合成的链含有一个随机多肽库,它们的 3'末端和 5‘末端都加了一个特定的侧翼序列,作为引物结合的位点,并且含有限制性内切酶的识别位点,以方便将文库 DNA 克隆进合适的载体系统。侧翼序列长度至少应该有 15bp,并且设计时应该有 3 个标准:1.它们应该是有效的 PCR 引物结合位点;2.它们应该含有后续克隆步骤所需的限制性酶切位点(可选:限制性酶切位点可以位于 PCR 引物 5’端与文库寡核苷酸的重叠区,而不要位于文库寡核苷酸内部),3.因为一端或两端的便翼 DNA 序列有可能在肽库中代表氨基酸,它们应该不包含影响文库功能的密码子。在合成文库的编码区过程中,每个密码子的前两个位点中,4 种核苷酸的含量是相等的,而在第三个位点,G+T 的比例是 1:1[图 26-1(a)]。在第三个位点不使用 A 和C 核苷酸,减少了形成终止密码子的可能,但仍允许编码 20 种氨基酸。
dNTP(各 20 mmol/L)
4. 其他
琼脂糖凝胶电泳所需的试剂和设备,包括溴化乙锭 (见第 11 步)
QIAprep Miniprep silica column(QIAGEN)
链霉抗生物素蛋白磁性珠(Dynal)
二、方法
1. 第一轮聚合反应:文库的 PCR 扩增
(1)进行下列反应,将单链的合成寡核苷酸,扩增为有效的构建文库的起始材料。下面所给的是 100ul 体系的用量。要构建大规模的库,准备 1 ml 的反应物,并将它们平分到 10个独立的 PCR 管中。
10pmol 的文库寡核苷酸
2U 的 Vent DNA 聚合酶
生物素标记的 PCR 引物,各 100pmol
10ul 的 10XVent 缓冲液
dATP、dCTP、dTTP 和 dGTP,各 1ul
用 ddH2O 定容至 100ul
(2)在一个热循环仪中,94°C 将混合物预热 20s。
(3)按下面的参数进行 30 轮 PCR 循环。
94°C10s
55°C10s(或用引物的合适退火温度)
72°C15s
(4)冷却至 4°C。
2. 第二轮聚合反应:合成配对的双链文库 DNA.
(5)在每 100ul 第一轮反应产物中,加入 10ul 的 10X 反应缓冲液、100pmol 的各引物、2U 的 Vent DNA 聚合酶。
(6)进行一个单轮的循环:94°C 30s,55°C 30s,72°C 15s。
(7)冷却至 4°C。
(8)用 QIAprep 的柱子纯化第二轮扩增产物,并用 10mmol/L Tris(pH8.0) 洗脱。
(9)用合适的内切酶消化洗脱产物(图 26-2,第 1 泳道),然后用乙醇沉淀 DNA, 并用ddH2O 溶解(Sambrook and Russell2001)。经过这一消化反应,生物素标记的 DNA 末端将从文库的核心释放出来。
(10)用链霉抗生物素蛋白磁性珠去掉生物素标记的 DNA 末端(Korn et al.1992)。这一步骤将增加目的连接产物的产量,因为这些末端能够与文库插入片段或表达载体相连接。链霉抗生物素蛋白的亲和步骤,同时能去掉任何没有酶切或部分酶切的 PCR 产物(图 26-2,第 3 泳道和第 4 泳道)。
(11)在 4% 的琼脂糖凝胶上,分析反应中间产物和终产物。终产物(图 26-2, 第 4 泳道)是高纯度且高度复杂的双链文库 DNA,有突出的末端,可以直接连接到合适的表达系统上。






