用 PCR 控制基因组 DNA 文库的质量实验
材料与仪器ddH20 Vent 缓冲液 Vent DNA 聚合酶 dNTP 质粒 DNA PCR 引物琼脂糖凝胶电泳所需的试剂和设备 热循环仪步骤一、材料1.
材料与仪器
ddH20 Vent 缓冲液 Vent DNA 聚合酶 dNTP 质粒 DNA PCR 引物
琼脂糖凝胶电泳所需的试剂和设备 热循环仪
琼脂糖凝胶电泳所需的试剂和设备 热循环仪
步骤
一、材料
1. 缓冲液、溶液和试剂
ddH20
2. 酶和酶缓冲液
10X Vent 缓冲液
Vent DNA 聚合酶
3. 核酸和寡核苷酸
dNTP,各 20 mmol/L
PCR 引物,可以与基因组 DNA 文库克隆位点两侧的载体序列退火
质粒 DNA(见下面第 1 步)
4. 特殊设备
琼脂糖凝胶电泳所需的试剂和设备,包括溴化乙锭 (见第 5 步)
热循环仪
二、方法
1. 在细菌中扩增后,用基因组文库制备的质粒 DNA 建立下列反应。
10~50ng 的质粒 DNA
50pmol 的各引物
5ul 的 10XVent 缓冲液
dATP、dCTP、dTTP 和 dGTP, 各 0.5ul
1U 的 Vent DNA 聚合酶
用 ddH20 定容至 50ul
对下面的每种质粒载体应该分别进行反应:
表达载体,没有文库 DNA 插入
基因组 DNA 文库的混合物
少量 DNA, 由基因组 DNA 文库的单个细菌克隆制备
2. 在一个热循环仪中,94°C 将混合物预热 20s。
3. 按下面的参数进行 30 轮 PCR 循环。
94°C15s
55°C10s(或用引物的合适退火温度)
72°C40s
将最后一次循环的延伸时间延长至 60s。
4. 冷却至 4C。
5. 取 25ul 的各反应产物,跑 4% 的琼脂糖凝胶电泳,并用溴化乙锭染色分析(Sambrook and Russell 2001)
三、分析
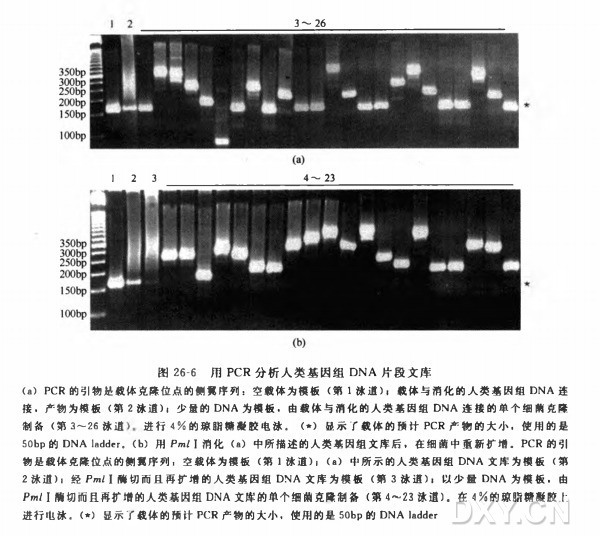
在图 26-6 所示的例子中,对文库混合物进行 PCR 分析,其中用的引物是 PmlⅠ位点的侧翼序列,结果显示,文库中含有目的大小的插入片段,但有大量的原始载体的污染 [图 26-6(a), 第 2 泳道]。从文库中选择单个克隆进行 PCR 分析,验证了用文库混合物做模板的结果,显示文库中的原始载体大约占 40%[所分析的 24 个克隆中有 10 个,图 26-6(a), 第 3~26 泳道]。为了提高文库的质量,用 PmlⅠ酶消化文库,然后在细菌中重新进行扩增,以减少原始载体的污染。对文库混合物进行 PCR 分析显示,用 PmlⅠ消化后,无插入的环状载体的污染大大减少了 [图 26-6(b), 第 2 泳道和第 3 泳道]。用 PmlⅠ消化又重新在细菌中扩增后,在文库中随机选择单个克隆进行 PCR 分析,证实无插入 DNA 的出现频率比原始文库大大降低 [20 个克隆中有 0 个,小于 5%,图 26-6(b), 第 4~23 泳道],并且克隆中含有预计大小的插入片段。对基因组片段文库中的克隆进行 DNA 序列分析显示,文库中含有预计大小的人类基因组 DNA 片段(数据没有列出)。因此,PCR 提供了一个快速、敏感和方便的方法,来分析原始的和改善过的文库。
1. 缓冲液、溶液和试剂
ddH20
2. 酶和酶缓冲液
10X Vent 缓冲液
Vent DNA 聚合酶
3. 核酸和寡核苷酸
dNTP,各 20 mmol/L
PCR 引物,可以与基因组 DNA 文库克隆位点两侧的载体序列退火
质粒 DNA(见下面第 1 步)
4. 特殊设备
琼脂糖凝胶电泳所需的试剂和设备,包括溴化乙锭 (见第 5 步)
热循环仪
二、方法
1. 在细菌中扩增后,用基因组文库制备的质粒 DNA 建立下列反应。
10~50ng 的质粒 DNA
50pmol 的各引物
5ul 的 10XVent 缓冲液
dATP、dCTP、dTTP 和 dGTP, 各 0.5ul
1U 的 Vent DNA 聚合酶
用 ddH20 定容至 50ul
对下面的每种质粒载体应该分别进行反应:
表达载体,没有文库 DNA 插入
基因组 DNA 文库的混合物
少量 DNA, 由基因组 DNA 文库的单个细菌克隆制备
2. 在一个热循环仪中,94°C 将混合物预热 20s。
3. 按下面的参数进行 30 轮 PCR 循环。
94°C15s
55°C10s(或用引物的合适退火温度)
72°C40s
将最后一次循环的延伸时间延长至 60s。
4. 冷却至 4C。
5. 取 25ul 的各反应产物,跑 4% 的琼脂糖凝胶电泳,并用溴化乙锭染色分析(Sambrook and Russell 2001)
三、分析
在图 26-6 所示的例子中,对文库混合物进行 PCR 分析,其中用的引物是 PmlⅠ位点的侧翼序列,结果显示,文库中含有目的大小的插入片段,但有大量的原始载体的污染 [图 26-6(a), 第 2 泳道]。从文库中选择单个克隆进行 PCR 分析,验证了用文库混合物做模板的结果,显示文库中的原始载体大约占 40%[所分析的 24 个克隆中有 10 个,图 26-6(a), 第 3~26 泳道]。为了提高文库的质量,用 PmlⅠ酶消化文库,然后在细菌中重新进行扩增,以减少原始载体的污染。对文库混合物进行 PCR 分析显示,用 PmlⅠ消化后,无插入的环状载体的污染大大减少了 [图 26-6(b), 第 2 泳道和第 3 泳道]。用 PmlⅠ消化又重新在细菌中扩增后,在文库中随机选择单个克隆进行 PCR 分析,证实无插入 DNA 的出现频率比原始文库大大降低 [20 个克隆中有 0 个,小于 5%,图 26-6(b), 第 4~23 泳道],并且克隆中含有预计大小的插入片段。对基因组片段文库中的克隆进行 DNA 序列分析显示,文库中含有预计大小的人类基因组 DNA 片段(数据没有列出)。因此,PCR 提供了一个快速、敏感和方便的方法,来分析原始的和改善过的文库。

相关文章
- PCR-SSCP的发展现状
- PCR-SSCP注意的事项
- PCR-SSCP原理和操作步骤
- 内切酶在PCR反应产物中的活性(Activity of Restriction Enzymes in a Primer Extensio
- RT-PCR Analysis--详细的RT-PCR方法
- RT-PCR: The Basics
- Real-Time or Kinetic PCR
- PCR Amplification from Microbial Colonies
- Calculating Concentrations for PCR
- PURIFICATION OF PCR PRODUCTS WITH SEPHAD